Dynamique des infections cellulaires

Cheffe d'équipe émergente
Pour survivre et se répliquer à l’intérieur d’une cellule, les bactéries, virus ou parasites intracellulaires doivent s’adapter à l’environnement intracellulaire afin de satisfaire leurs besoins. En réponse à cette invasion, la cellule hôte met en place des mécanismes de défense pour éliminer le pathogène. Nos recherches portent sur ces interactions hautement dynamiques entre les pathogènes intracellulaires et leurs cellules hôtes.
Les pathogènes intracellulaires ont évolué pour devenir des enseignants involontaires de la biologie cellulaire. Par leur perturbation spécifique de la biologie cellulaire normale, ils nous révèlent des aspects nouveaux des mécanismes moléculaires qui contrôlent les événements cellulaires dynamiques.

Notre équipe de recherche émergente vise à comprendre les dynamiques moléculaires qui se déploient au cours des infections intracellulaires. Notre principal pathogène d’intérêt est Salmonella, une bactérie d’origine alimentaire classée comme « prioritaire » par l’Organisation mondiale de la santé et un modèle polyvalent pour l’étude de la pathogenèse bactérienne intracellulaire.
Nous étudions comment Salmonella détourne les voies de signalisation de la cellule hôte afin de remodeler l’architecture intracellulaire et d’établir une niche unique permissive à la réplication, qui fournit des nutriments tout en limitant la détection par l’hôte. Un axe central de nos travaux porte sur la manière dont les modifications post-traductionnelles remodèlent le trafic intracellulaire au cours de l’infection, conduisant finalement à la formation d’une vacuole spécialisée contenant la bactérie et répondant à ses besoins.
Comme la réplication intravacuolaire est une stratégie partagée par des pathogènes humains majeurs, notamment Mycobacterium tuberculosis, Chlamydia trachomatis et Legionella pneumophila, nos recherches offrent un cadre pour identifier des principes communs de formation de niches. En mettant au jour ces mécanismes partagés, nous visons à orienter le développement de nouvelles stratégies thérapeutiques ciblant les refuges intracellulaires dont dépendent les pathogènes pour survivre.
Équipe
Chercheuse
Virginie Stévenin
Master Student
Esteban Francois
Stévenin et al. (2024) Multi-omic analyses of cancer-associated clinical Salmonella reveal a bacterial-induced host metabolic shift leading to cell transformation. Cell Rep
Stévenin & Neefjes (2022) Control of host PTMs by intracellular bacteria: an opportunity towards novel anti-infective agents. Cell Chem Biol
Stévenin et al. (2021) Purification of infection-associated macropinosomes by magnetic isolation for proteomic characterization. Nat Protoc
Stévenin et al. (2019) Dynamic growth and shrinkage of the Salmonella-containing vacuole determines the intracellular pathogen niche. Cell Rep
Voznica et al. (2018). Identifying parameters of host cell vulnerability during Salmonella infection by quantitative image analysis and modeling. Infect Immun
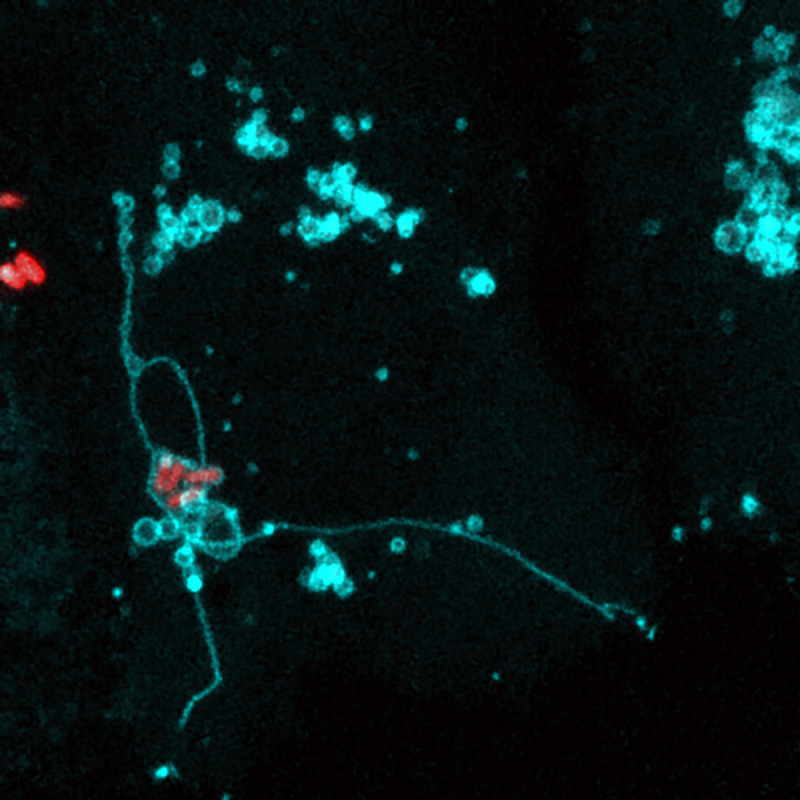
Salmonella building a vacuolar niche in an epithelial cell.
2026
Paradis M#, Gorse L#, Silva Da Costa L, Chua R, Alberola G, Metais A, Batistic L, Benoist T, David A, Pages E, Gomes A, Bomfim C, Ravon-Katossky L, Suire B, Fromont L, Siaden-Ortega R, Pericat D, Cougoule C, Boyer L, Gaudenzio N, Mazars R, Stévenin V, Zhong F, Olagnier D, Meunier E*. (2026) A TAK1-driven NLRP1 inflammasome pathway revealed by phosphatase-targeting environmental toxins. BioRxiv doi:10.64898/2026.01.23.701233
Sapmaz A, van der Zanden S, Akkermans J, Menager L, Janssen L, Francois E, Hagoort N, Berlin I, Neefjes J, Stévenin V*. (2026) Salmonella exploits USP32 to coordinate Rab14 and Rab11 recycling pathways for intracellular survival. BioRxiv doi:10.64898/2026.01.01.696821
2025
De Meyer M, Verhee A, Grzesik H, De Sutter D, Huyghe J, Delhaye L, Van de Steene T, Fijalkowski I, Jonckheere V, Meuris L, Bertrand M, Van Damme P#*, Stévenin V#*, Eyckerman S#*. (2025) Virotrap Reveals Salmonella SopB as A Ubiquitinated Cargo for Host ESCRT-0. BioRxiv doi:10.1101/2025.08.19.669813
Stévenin V, Neefjes J*. (2025) Salmonella, the insidious contributor to gallbladder and colon cancers. Nature Reviews Cancer. doi:10.1038/s41568-025-00812-z
2024
Stévenin V#*, Coipan C#, Duijster J, van Elsland D, Voogd L, Bigey L, van Hoek A, Wijnands L, Jansen L, Akkermans J, Neefjes-Borst A, Franz E, Mughini-Gras L, Neefjes J*. (2024) Multi-omics analyses of cancer-linked clinical salmonellae reveal bacterial-induced host metabolic shift and mTOR-dependent cell transformation. Cell Reports. 43(11):114931. doi:10.1016/j.celrep.2024.114931
2023
Stévenin V*, Neefjes J. (2023). Soft agar colony formation assay to quantify mouse embryonic fibroblast transformation after Salmonella infection. STAR Protocols. 4(3):102379. doi: 10.1016/j.xpro.2023.10237
Berlin I*, Sapmaz A, Stévenin V, Neefjes J. (2023) Ubiquitin and its relatives as wizards of the endolysosomal system. Journal of Cell Science. 136 (4): jcs260101. doi: 10.1242/jcs.260101
2022
van Elsland D#, Duijster J#, Zhang J#, Stévenin V, Zhang Y, Zha L, Xia Y, Franz E, Sun J*, Mughini-Gras L*, Neefjes J*. (2022) Repetitive non-typhoidal Salmonella exposure is an environmental risk factor for colon cancer and tumor growth. Cell Reports Medicine. 3: 100852. doi: 10.1016/j.xcrm.2022.100852
Stévenin V*, Neefjes J. (2022). Control of host PTMs by intracellular bacteria: an opportunity towards novel anti-infective agents. Cell Chemical Biology. 29(5):741-756. doi: 10.1016/j.chembiol.2022.04.004.
Schink K*, Tan KW, Spangenberg H, Martorana D, Sneeggen M, Stévenin V, Enninga J, Campsteijn C, Raiborg C, Stenmark H*. (2021) The phosphoinositide coincidence detector Phafin2 promotes macropinocytosis by coordinating actin organisation at forming macropinosomes. Nature Communications 12(1):6577. doi: 10.1038/s41467-021-26775-x
2021
Stévenin V*, Giai Gianetto Q, Duchateau M, Matondo M, Enninga J, Chang YY*. (2021) Purification of infection-associated macropinosomes by magnetic isolation for proteomic characterization. Nature Protocols 16(11):5220-5249. doi: 10.1038/s41596-021-00610-5
Chang YY*, Enninga J, Stévenin V*. (2021) New methods to decrypt emerging macropinosome functions during the host-pathogen crosstalk. Cellular Microbiology 23(7):e13342. doi: 10.1111/cmi.13342.
2020
Chang YY, Stévenin V, Duchateau M, Giai Gianetto Q, Hourdel V, Rodrigues CD, Matondo M, Reiling N, Enninga J*. (2020) Shigella hijacks the exocyst to cluster macropinosomes for efficient vacuolar escape. PLoS Pathogens. 16(8):e1008822. doi: 10.1371/journal.ppat.1008822.
2019
Stévenin V, Enninga J*. (2019) Cellular imaging of intracellular bacterial pathogens. Microbiology Spectrum 7(2). doi:10.1128/microbiolspec.BAI-0017-2019
Stévenin V, Chang YY, Le Toquin Y, Duchateau M, Gianetto QG, Luk CH, Salles A, Sohst V, Matondo M, Reiling N, Enninga J*. (2019) Dynamic growth and shrinkage of the Salmonella-containing vacuole determines the intracellular pathogen niche. Cell Reports. 29(12):3958-3973.e7. doi: 10.1016/j.celrep.2019.11.049.
2018
Voznica J, Enninga J, Stévenin V*. (2018) High-throughput microscopic analysis of Salmonella invasion of host cells. Bio-protocol 20(18):e3017. doi: 10.21769/BioProtoc.3017.
Fredlund J#, Santos JC#, Stévenin V#, Weiner A, Rechav K, Mallet A, Krijnse-Locker J, Elbaum M, Enninga J*. (2018) The early entry of Salmonella in a distinct tight compartment revealed at high temporal and ultrastructural resolution. Cellular Microbiology. 20(4):e12816. doi: 10.1111/cmi.12816.
Voznica J, Gardella C, Belotserkovsky I, Dufour A, Enninga J, Stévenin V*. (2018) Identifying parameters of host cell vulnerability during Salmonella infection by quantitative image analysis and modeling. Infection and Immunity. 86(1):e00644-17. doi: 10.1128/IAI.00644-17
- Collaborations
Étienne Meunier, IPBS, Toulouse, France
Agnès Wiedemann, IRSD, Toulouse, France
Pedro Escoll, Institut Pasteur, Paris, France
Jost Enninga, Institut Pasteur, Paris, France
Kristine Schauer, Institut Gustave Roussy, Paris, France
Étienne Morel, Institut Necker Enfants Malades, Paris, France
Ting-Di Wu, Institut Curie, Paris, France
Franck Perez, Institut Curie, Paris, France
Sven Eyckerman, VIB-Gand, Belgique
Petra van Damme, VIB-Gand, Belgique
Adán Pinto-Fernández, CAMS Oxford Institute, Royaume-Uni
Jacques Neefjes, LUMC, Leyde, Pays-Bas
Mikael Sellin, Université d’Uppsala, Suède
Financements
- Bourse VENI du programme Talent de la NWO (2025–2027)
- Programme « Amorçage de jeunes équipes » de la Fondation pour la Recherche Médicale (2026–2029)


