Compréhension et ciblage de la réparation de l’ADN

Responsable d'équipe
Les recherches de l’équipe « Compréhension et Ciblage de la Réparation de l’ADN » (DDR lab) visent à découvrir comment les cellules humaines répondent aux dommages de l’ADN afin d’identifier et de valider de nouvelles cibles thérapeutiques pour traiter le cancer. Nos découvertes majeures incluent la découverte d’une voie de réparation alternative des cassures de l’ADN (2004), des mécanismes éliminant la protéine Ku sur certaines cassures (2016, 2020), d’une nouvelle fonction de la protéine BRCA1 aux centromères (2014, 2021), des mécanismes d’assemblage des protéines de réparation sur le détecteur Ku (2018, 2023) et du mécanisme d’action de plusieurs petites molécules d’intérêt thérapeutique (2017, 2021, 2022).
A travers une approche multidisciplinaire, nous cherchons à résoudre une question importante : comment fonctionne la réparation de l’ADN et comment peut-on la moduler à l’aide de petites molécules ?

La réparation de l’ADN détermine les effets de nombreuses thérapies anticancéreuses. Ainsi, il y a un intérêt considérable pour identifier de nouvelles petites molécules capables de moduler la réparation de l’ADN. Dans l’équipe DDR, nous cherchons à comprendre comment les cellules répondent aux dommages de l’ADN, plus particulièrement au type de dommages le plus toxique, les Cassures Double-Brin de l’ADN (CDBs). Nous utilisons ensuite nos découvertes fondamentales pour développer et conduire des cribles ciblés et phénotypiques pour identifier des modulateurs d’étapes spécifiques de la réponse aux dommages de l’ADN. Ces modulateurs pourraient constituer de nouveaux candidats médicaments pour améliorer les thérapies anticancéreuses. Pour atteindre ces objectifs, nous utilisons la microscopie à haute et super-résolution, des techniques de pointes pour induire des dommages de l’ADN dans des positions précises du noyau ou du génome, des approches de biologie cellulaire et moléculaire, des analyses génétiques et génomiques, des cribles de petites molécules et des sondes chimiques. Nous bénéficions de multiples collaborations avec des équipes de chimistes et de biologie structurale. Nous avons récemment implémenté dans l’équipe plusieurs techniques complémentaires qui nous ont permis de découvrir le mécanismes d’action de petites molécules d’intérêt pour la lutte contre le cancer (Bombarde et al. Mol Cancer Ther 2017; Bossaert, Pipier et al. 2021; Demange, Joly, Marcoux et al. eLife 2022).
Les relations entre les différentes voies de réparation de l’ADN
Deux mécanismes principaux de réparation des CDBs co-existent dans les cellules humaines : la Jonction d’Extrémités Non-Homologues (JENH) et la Recombinaison Homologue (RH). Le complexe Ku fait l’objet d’un intérêt plus particulier dans l’équipe car il constitue le détecteur majeur des CDBs chez l’homme dont il initie la réparation par JENH. Par le développement d’une nouvelle méthode permettant de visualiser et de quantifier l’accumulation des protéines de JENH au niveau de virtuellement toute CDB (Britton et al. J Cell Biol 2013), nous avons récemment découvert les mécanismes permettant d’éliminer les protéines de JENH de l’extrémité des CDB qui doivent être réparées par RH. Nous avons montré que ces mécanismes peuvent être contrôlés par des petites molécules et que leur inhibition induit des événements de réparation toxiques en réponse à certains agents anticancéreux (Britton et al. 2013 J Cell Biol; Chanut, Britton et al. 2016 Nat Commun; Britton, Chanut et al. 2020 Nucleic Acids Res).
Comment les protéines de réparation s’assemblent sur la chromatine endommagée
Grace à nos collaborations avec des biologistes structuraux (J.-B.Charbonnier, I2BC, Paris ; A. Chaplin, University of Leicester, UK), nous avons caractérisé comment les protéines de réparation APLF, XLF et plus récemment PAXX s’associent avec le détecteur majeur des CDBs chez l’homme, le complexe Ku. Les interfaces spécifiques sur Ku et les domaines de ces protéines impliqués dans ces interactions ont été caractérisés par résolution de la structure de ces complexes par CryoEM et cristallographie aux rayons X et par réalisation de mutants (Nemoz, Ropars, Frit et al. 2018 Nat Struct Mol Biol; Seif-El-Dahan, Kefala-Stavridi, Frit, Hardwick et al. 2023). Une technique de pointe permettant d’utiliser un laser bi-photon pour réaliser des dommages dans des régions du noyau de cellules humaines a permis de suivre en temps réel la dynamique d’assemblage des protéines et de leurs mutants permettant une analyse de leurs effets fonctionnels. Ces nouvelles interfaces peuvent maintenant faire l’objet de crible pour bloquer l’assemblage de protéines spécifiques sur Ku. Cette technique a également permis d’analyser comment le contexte génomique, comme l’hétérochromatine ou la transcription influe sur la réponse aux dommages de l’ADN. Ainsi, nous avons découverts un mécanisme permettant l’exclusion de la chromatine endommagée d’un grand nombre de facteurs se liant aux ARN, permettant ainsi de limiter la formation locale de R-loops (Britton, Dernoncourt et al. Nucleic Acids Res 2014).
Comment la stabilisation de structures non-B de l’ADN induit des cassures de l’ADN
Certaines petites molécules stabilisant des structures secondaires de l’ADN, comme les ligands de G-quadruplexes (G4), induisent des dommages de l’ADN dont des CDBs (Zell et al. 2020 RSC Chem Biol). A travers une approche génomique non biaisée, nous avons récemment découvert que l’ADN topoisomérase 2 alpha (TOP2A) est responsable de la production des CDBs induites par plusieurs ligands de G4 dont le CX-5461, une petite molécule actuellement en essais cliniques en oncologie (Bossaert, Pipier et al. 2021 eLife). En collaboration avec les équipes de D. Monchaud (ICMUB, Dijon) et A. Granzhan (Institut Curie, Paris), nous avons également contribué à développer de nouveaux ligands d’une autre structure secondaire de l’ADN, les Jonctions de l’ADN à Trois Voies (TWJ) et montré que ces molécules induisent également des dommages de l’ADN par un mécanisme que nous sommes actuellement en train de caractériser (Duskova 2019 J Med Chem; Duskova et al. 2020 J Am Chem Soc; Zell et al. Nucleic Acids Res 2021)
Team members
Chercheurs
Nadia Barboule (CNRS)
Sébastien Britton (CNRS)
Patrick Calsou (Inserm)
Philippe Frit (CNRS)
Dennis Gomez (CNRS)
Léa Marie (University)
Florence Larminat (CNRS)
Marie-Jeanne Pillaire (Inserm)
Ingénieurs et Techniciens
Antonio Peixoto (CNRS)
Nadège Preuilh
Amandine Prioux-Quartier
Carine Racca (CNRS)
Doctorants
Maëlle Caroff
Jeanne Chauvat
Bastien Dumais
Nos projets de recherche
Pour en savoir plus sur nos différents projets de recherche vous pouvez consulter la version anglaise du site.
Demange*, Joly*, Marcoux* et al. (2022) SDR enzymes oxidize specific lipidic alkynylcarbinols into cytotoxic protein-reactive species. eLife
Bossaert*, Pipier* et al. (2021) Transcription-associated topoisomerase 2alpha (TOP2A) activity is a major effector of cytotoxicity induced by G-quadruplex ligands. eLife
Racca et al. (2021) BRCA1 prevents R-loop-associated centromeric instability. Cell Death Dis
Britton*, Chanut* et al. (2020) ATM antagonizes NHEJ proteins assembly and DNA-ends synapsis at single-ended DNA double strand breaks. Nucleic Acids Res
Zell et al. (2021) Dual targeting of higher-order DNA structures by azacryptands induces DNA junction-mediated DNA damage in cancer cells. Nucleic Acids Res
Nemoz*, Ropars*, Frit* et al. (2018) XLF and APLF bind Ku80 at two remote sites to ensure DNA repair by non-homologous end joining. Nat Struct Mol Biol
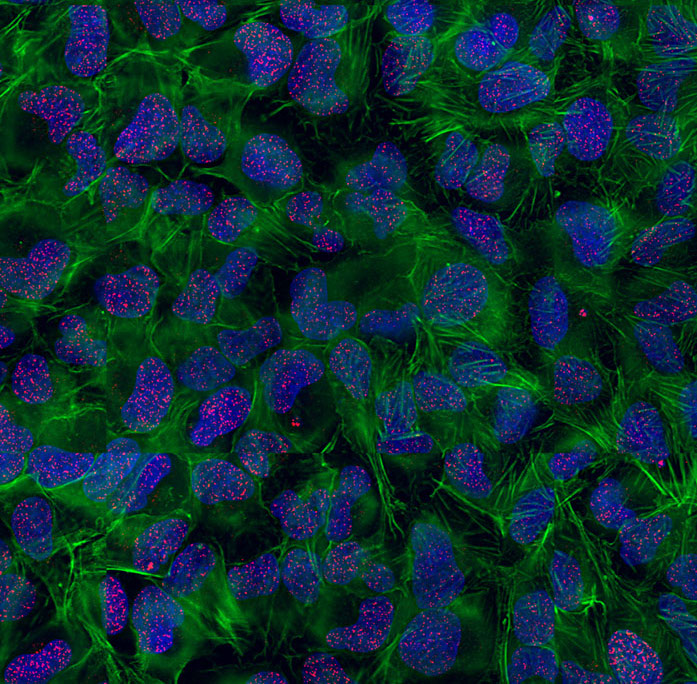
Image à haute résolution des Cassures Double-Brin de l’ADN (CDBs) dans des cellules humaines traitées par des radiations ionisantes en présence d’un inhibiteur de la réponse aux dommages de l’ADN. Rouge: CDBs visualisées par gammaH2AX; Vert: cytosquelette d’Actine; Bleu : ADN.
© Sébastien Britton
The complete list of our publications is available through Pubmed
Publications
* incidates (co-) corresponding authors ; # indicates equal contribution
2023
Pinto, L.M.#, Pailas, A. #, Bondarchenko, M., Sharma, A.B., Neumann, K., Rizzo, A.J., Jeanty, C., Nicot, N., Racca, C., Graham, M.K., Naughton, C., Liu, Y., Chen, C.L., Meakin, P.J., Gilbert, N., Britton, S., Meeker, A.K., Heaphy, C.M., Larminat, F.*, Van Dyck, E.* (2023) DAXX promotes centromeric stability independently of ATRX by preventing the accumulation of R-loop-induced DNA double-stranded breaks. Nucleic Acids Res (View in Nucleic Acids Research)
Kefala Stavridi, A.#, Gontier, A. #, Morin, V. #, Frit, P. #, Ropars, V., Barboule, N., Racca, C., Jonchhe, S., Morten, M.J., Andreani, J., Rak, A., Legrand, P., Bourand-Plantefol, A., Hardwick, S.W., Chirgadze, D.Y., Davey, P., De Oliveira, T.M., Rothenberg, E., Britton, S.*, Calsou, P.*, Blundell, T.L.*, Varela, P.F.*, Chaplin, A.K.*, Charbonnier, J.B.* (2023) Structural and functional basis of inositol hexaphosphate stimulation of NHEJ through stabilization of Ku-XLF interaction. Nucleic Acids Res 51, 11732-11747 (View in Nucleic Acids Research)
Bossuat, M., Rulliere, P., Preuilh, N., Peixoto, A., Joly, E., Gomez, J.G., Bourkhis, M., Rodriguez, F., Goncalves, F., Fabing, I., Gaspard, H., Bernardes-Genisson, V., Maraval, V., Ballereau, S., Chauvin, R.*, Britton, S.*, Genisson, Y.* (2023) Phenyl dialkynylcarbinols, a Bioinspired Series of Synthetic Antitumor Acetylenic Lipids. J Med Chem 66, 13918-13945 (View in the Journal of Medicinal Chemistry)
Chebaiki, M., Delfourne, E., Tamhaev, R., Danoun, S., Rodriguez, F., Hoffmann, P., Grosjean, E., Goncalves, F., Azema-Despeyroux, J., Pal, A., Kordulakova, J., Preuilh, N., Britton, S., Constant, P., Marrakchi, H., Maveyraud, L.*, Mourey, L.*, Lherbet, C.* (2023) Discovery of new diaryl ether inhibitors against Mycobacterium tuberculosis targeting the minor portal of InhA. European journal of medicinal chemistry 259, 115646 (View in the European Journal of Medicinal Chemistry)
Seif-El-Dahan, M. #, Kefala-Stavridi, A. #, Frit, P. #, Hardwick, S.W. #, Chirgadze, D.Y., Maia De Oliviera, T., Andreani, J., Britton, S., Barboule, N., Bossaert, M., Pandurangan, A.P., Meek, K., Blundell, T.L., Ropars, V.*, Calsou, P.*, Charbonnier, J.B.*, Chaplin, A.K.* (2023) PAXX binding to the NHEJ machinery explains functional redundancy with XLF. Sci Adv 9, eadg2834 (View in Science Advances)
2022
Demange, P.#, Joly, E. #, Marcoux, J. #, Zanon, P.R.A., Listunov, D., Rulliere, P., Barthes, C., Noirot, C., Izquierdo, J.B., Rozie, A., Pradines, K., Hee, R., de Brito, M.V., Marcellin, M., Serre, R.F., Bouchez, O., Burlet-Schiltz, O., Oliveira, M.C.F., Ballereau, S., Bernardes-Genisson, V., Maraval, V., Calsou, P., Hacker, S.M., Genisson, Y.*, Chauvin, R.*, Britton, S.* (2022) SDR enzymes oxidize specific lipidic alkynylcarbinols into cytotoxic protein-reactive species. eLife 11 (View in eLife)
2021
Racca, C., Britton, S., Hedouin, S., Francastel, C., Calsou, P., Larminat, F.* (2021) BRCA1 prevents R-loop-associated centromeric instability. Cell death & disease 12, 896 (View in Cell Death & Disease)
Zell, J., Duskova, K., Chouh, L., Bossaert, M., Cheron, N., Granzhan, A., Britton, S.*, Monchaud, D.* (2021) Dual targeting of higher-order DNA structures by azacryptands induces DNA junction-mediated DNA damage in cancer cells. Nucleic Acids Res 49, 10275-10288 (View in Nucleic Acids Research)
Sharma, A.B., Erasimus, H., Pinto, L., Caron, M.C., Gopaul, D., Peterlini, T., Neumann, K., Nazarov, P.V., Fritah, S., Klink, B., Herold-Mende, C.C., Niclou, S.P., Pasero, P., Calsou, P., Masson, J.Y., Britton, S., Van Dyck, E. (2021) XAB2 promotes Ku eviction from single-ended DNA double-strand breaks independently of the ATM kinase. Nucleic Acids Res 49, 9906-9925 (View in Nucleic Acids Research)
Pipier, A., Devaux, A., Lavergne, T., Adrait, A., Coute, Y., Britton, S., Calsou, P., Riou, J.F., Defrancq, E., Gomez, D.* (2021) Constrained G4 structures unveil topology specificity of known and new G4 binding proteins. Scientific reports 11, 13469 (View in Scientific reports)
Bossaert, M.#, Pipier, A. #, Riou, J.F., Noirot, C., Nguyen, L.T., Serre, R.F., Bouchez, O., Defrancq, E., Calsou, P.*, Britton, S.*, Gomez, D.* (2021) Transcription-associated topoisomerase 2alpha (TOP2A) activity is a major effector of cytotoxicity induced by G-quadruplex ligands. eLife 10 (View in eLife)
2020
Zell, J., Rota Sperti, F., Britton, S.*, Monchaud, D.* (2021) DNA folds threaten genetic stability and can be leveraged for chemotherapy. RSC Chem Biol 2, 47-76 (View this review in RSC Chemical Biology)
Britton, S.*,#, Chanut, P. #, Delteil, C., Barboule, N., Frit, P., Calsou, P.* (2020) ATM antagonizes NHEJ proteins assembly and DNA-ends synapsis at single-ended DNA double strand breaks. Nucleic Acids Res 48, 9710-9723 (View in Nucleic Acids Research)
Janel-Bintz, R., Kuhn, L., Frit, P., Chicher, J., Wagner, J., Haracska, L., Hammann, P., Cordonnier, A.M.* (2020) Proteomic Analysis of DNA Synthesis on a Structured DNA Template in Human Cellular Extracts: Interplay Between NHEJ and Replication-Associated Proteins. Proteomics 20, e1900184 (View in Proteomics)
Duskova, K., Lejault, P., Benchimol, E., Guillot, R., Britton, S.*, Granzhan, A.*, Monchaud, D.* (2020) DNA Junction Ligands Trigger DNA Damage and Are Synthetic Lethal with DNA Repair Inhibitors in Cancer Cells. J Am Chem Soc 142, 424-435 (View in the Journal of the American Chemical Society)
2019
Cristini, A., Ricci, G., Britton, S., Salimbeni, S., Huang, S.N., Marinello, J., Calsou, P., Pommier, Y., Favre, G., Capranico, G., Gromak, N.*, Sordet, O.* (2019) Dual Processing of R-Loops and Topoisomerase I Induces Transcription-Dependent DNA Double-Strand Breaks. Cell reports 28, 3167-3181 e3166 (View in Cell Reports)
David, A.P., Pipier, A., Pascutti, F., Binolfi, A., Weiner, A.M.J., Challier, E., Heckel, S., Calsou, P., Gomez, D., Calcaterra, N.B., Armas, P.* (2019) CNBP controls transcription by unfolding DNA G-quadruplex structures. Nucleic Acids Res 47, 7901-7913 (View in Nucleic Acids Research)
Duskova, K., Lamarche, J., Amor, S., Caron, C., Queyriaux, N., Gaschard, M., Penouilh, M.J., de Robillard, G., Delmas, D., Devillers, C.H., Granzhan, A., Teulade-Fichou, M.P., Chavarot-Kerlidou, M., Therrien, B., Britton, S., Monchaud, D.* (2019) Identification of Three-Way DNA Junction Ligands through Screening of Chemical Libraries and Validation by Complementary in Vitro Assays. J Med Chem 62, 4456-4466 (View in the Journal of Medicinal Chemistry)
Pipier, A., De Rache, A., Modeste, C., Amrane, S., Mothes-Martin, E., Stigliani, J.L., Calsou, P., Mergny, J.L., Pratviel, G., Gomez, D. (2019) G-Quadruplex binding optimization by gold(iii) insertion into the center of a porphyrin. Dalton Trans 48, 6091-6099 (View in Dalton transactions)
Frit, P., Ropars, V., Modesti, M., Charbonnier, J.B.*, Calsou, P.* (2019) Plugged into the Ku-DNA hub: The NHEJ network. Progress in biophysics and molecular biology 147, 62-76 (View this review in Progress in Biophysics and Molecular Biology)
2018
Rozie, A., Santos, C., Fabing, I., Calsou, P., Britton, S.*, Genisson, Y., Ballereau, S.* (2018) Alkyne-Tagged Analogue of Jaspine B: New Tool for Identifying Jaspine B Mode of Action. Chembiochem 19, 2438-2442 (View in ChemBioChem)
Nemoz, C.#, Ropars, V. #, Frit, P. #, Gontier, A., Drevet, P., Yu, J., Guerois, R., Pitois, A., Comte, A., Delteil, C., Barboule, N., Legrand, P., Baconnais, S., Yin, Y., Tadi, S., Barbet-Massin, E., Berger, I., Le Cam, E., Modesti, M., Rothenberg, E., Calsou, P.*, Charbonnier, J.B.* (2018) XLF and APLF bind Ku80 at two remote sites to ensure DNA repair by non-homologous end joining. Nat Struct Mol Biol 25, 971-980 (View in Nature Structural & Molecular Biology)
2017
Bombarde, O., Larminat, F., Gomez, D., Frit, P., Racca, C., Gomes, B., Guilbaud, N., Calsou, P.* (2017) The DNA-Binding Polyamine Moiety in the Vectorized DNA Topoisomerase II Inhibitor F14512 Alters Reparability of the Consequent Enzyme-Linked DNA Double-Strand Breaks. Mol Cancer Ther 16, 2166-2177 (View in Molecular Cancer Therapeutics)
Gueddouda, N.M., Mendoza, O., Gomez, D., Bourdoncle, A.*, Mergny, J.L.* (2017) G-quadruplexes unfolding by RHAU helicase. Biochim Biophys Acta Gen Subj 1861, 1382-1388 (View in Biochimica et Biophysica Acta (BBA) – General Subjects)
2016
Chanut, P. #, Britton, S.*,#, Coates, J., Jackson, S.P.*, Calsou, P.* (2016) Coordinated nuclease activities counteract Ku at single-ended DNA double-strand breaks. Nat Commun 7, 12889 (View in Nature communications)
Hegde, M.L., Dutta, A., Yang, C., Mantha, A.K., Hegde, P.M., Pandey, A., Sengupta, S., Yu, Y., Calsou, P., Chen, D., Lees-Miller, S.P., Mitra, S. (2016) Scaffold attachment factor A (SAF-A) and Ku temporally regulate repair of radiation-induced clustered genome lesions. Oncotarget 7, 54430-54444 (View in Oncotarget)
Lamaa, A., Le Bras, M., Skuli, N., Britton, S., Frit, P., Calsou, P., Prats, H., Cammas, A., Millevoi, S.* (2016) A novel cytoprotective function for the DNA repair protein Ku in regulating p53 mRNA translation and function. EMBO Rep 17, 508-518 (View in EMBO reports)
Menchon, G., Bombarde, O., Trivedi, M., Negrel, A., Inard, C., Giudetti, B., Baltas, M., Milon, A., Modesti, M., Czaplicki, G., Calsou, P. (2016) Structure-Based Virtual Ligand Screening on the XRCC4/DNA Ligase IV Interface. Scientific reports 6, 22878 (View in Scientific reports)
Chabalier-Taste, C., Brichese, L., Racca, C., Canitrot, Y., Calsou, P., Larminat, F.* (2016) Polo-like kinase 1 mediates BRCA1 phosphorylation and recruitment at DNA double-strand breaks. Oncotarget 7, 2269-2283 (View in Oncotarget)
2015
Yuan, Y. #, Britton, S. #, Delteil, C., Coates, J., Jackson, S.P., Barboule, N., Frit, P., Calsou, P.* (2015) Single-stranded DNA oligomers stimulate error-prone alternative repair of DNA double-strand breaks through hijacking Ku protein. Nucleic Acids Res 43, 10264-10276 (View in Nucleic Acids Research)
Brown, J.S. #, Lukashchuk, N. #, Sczaniecka-Clift, M.†, Britton, S. †, le Sage, C., Calsou, P., Beli, P.*, Galanty, Y.*, Jackson, S.P.* (2015) Neddylation promotes ubiquitylation and release of Ku from DNA-damage sites. Cell reports 11, 704-714 (View in Cell reports)
Sabater, L., Nicolau-Travers, M.L., De Rache, A., Prado, E., Dejeu, J., Bombarde, O., Lacroix, J., Calsou, P., Defrancq, E., Mergny, J.L., Gomez, D., Pratviel, G.* (2015) The nickel(II) complex of guanidinium phenyl porphyrin, a specific G-quadruplex ligand, targets telomeres and leads to POT1 mislocalization in culture cells. J Biol Inorg Chem 20, 729-738 (View in the Journal of Biological Inorganic Chemistry)
Douglas, P., Ye, R., Morrice, N., Britton, S., Trinkle-Mulcahy, L., Lees-Miller, S.P.* (2015) Phosphorylation of SAF-A/hnRNP-U Serine 59 by Polo-Like Kinase 1 Is Required for Mitosis. Mol Cell Biol 35, 2699-2713 (View in Molecular and Cellular Biology)
2014
Di Paolo, A., Racca, C., Calsou, P., Larminat, F.* (2014) Loss of BRCA1 impairs centromeric cohesion and triggers chromosomal instability. FASEB J 28, 5250-5261 (View in the FASEB Journal)
Britton, S. #, Dernoncourt, E. #, Delteil, C., Froment, C., Schiltz, O., Salles, B., Frit, P., Calsou, P.* (2014) DNA damage triggers SAF-A and RNA biogenesis factors exclusion from chromatin coupled to R-loops removal. Nucleic Acids Res 42, 9047-9062 (View in Nucleic Acids Research)
Larrieu, D., Britton, S., Demir, M., Rodriguez, R.*, Jackson, S.P.* (2014) Chemical inhibition of NAT10 corrects defects of laminopathic cells. Science 344, 527-532 (View in Science)
Frit, P., Barboule, N., Yuan, Y., Gomez, D., Calsou, P. (2014) Alternative end-joining pathway(s): bricolage at DNA breaks. DNA Repair (Amst) 17, 81-97 (View in DNA repair)
Abet, V., Mariani, A., Truscott, F.R., Britton, S., Rodriguez, R. (2014) Biased and unbiased strategies to identify biologically active small molecules. Bioorg Med Chem 22, 4474-4489 (View in Bioorganic & Medicinal Chemistry)
2013
Britton, S., Coates, J., Jackson, S.P.* (2013) A new method for high-resolution imaging of Ku foci to decipher mechanisms of DNA double-strand break repair. J Cell Biol 202, 579-595 (View in the Journal of Cell Biology)
Cottarel, J., Frit, P., Bombarde, O., Salles, B., Negrel, A., Bernard, S., Jeggo, P.A., Lieber, M.R., Modesti, M., Calsou, P.* (2013) A noncatalytic function of the ligation complex during nonhomologous end joining. J Cell Biol 200, 173-186 (View in the Journal of Cell Biology)
Fallone, F., Britton, S., Nieto, L., Salles, B., Muller, C. (2013) ATR controls cellular adaptation to hypoxia through positive regulation of hypoxia-inducible factor 1 (HIF-1) expression. Oncogene 32, 4387-4396 (View in Oncogene)
2012
Rodriguez, R.#, Miller, K.M. #, Forment, J.V. #, Bradshaw, C.R., Nikan, M., Britton, S., Oelschlaegel, T., Xhemalce, B., Balasubramanian, S.*, Jackson, S.P.* (2012) Small-molecule-induced DNA damage identifies alternative DNA structures in human genes. Nat Chem Biol 8, 301-310 (View in Nature Chemical Biology)
2011
Cheng, Q., Barboule, N., Frit, P., Gomez, D., Bombarde, O., Couderc, B., Ren, G.S.*, Salles, B.*, Calsou, P.* (2011) Ku counteracts mobilization of PARP1 and MRN in chromatin damaged with DNA double-strand breaks. Nucleic Acids Res 39, 9605-9619 (View in Nucleic Acids Research)
Romera, C., Bombarde, O., Bonnet, R., Gomez, D., Dumy, P., Calsou, P., Gwan, J.F., Lin, J.H., Defrancq, E., Pratviel, G.* (2011) Improvement of porphyrins for G-quadruplex DNA targeting. Biochimie 93, 1310-1317 (View in Biochimie)
2010
Miller, K.M.#, Tjeertes, J.V. #, Coates, J., Legube, G., Polo, S.E., Britton, S., Jackson, S.P.* (2010) Human HDAC1 and HDAC2 function in the DNA-damage response to promote DNA nonhomologous end-joining. Nat Struct Mol Biol 17, 1144-1151 (View in Nature Structural & Molecular Biology)
Gomez, D., Guedin, A., Mergny, J.L., Salles, B., Riou, J.F., Teulade-Fichou, M.P., Calsou, P.* (2010) A G-quadruplex structure within the 5′-UTR of TRF2 mRNA represses translation in human cells. Nucleic Acids Res 38, 7187-7198 (View in Nucleic Acids Research)
Bombarde, O., Boby, C., Gomez, D., Frit, P., Giraud-Panis, M.J., Gilson, E., Salles, B., Calsou, P.* (2010) TRF2/RAP1 and DNA-PK mediate a double protection against joining at telomeric ends. EMBO J 29, 1573-1584 (View in the EMBO Journal)
Ye, J.#, Lenain, C. #, Bauwens, S., Rizzo, A., Saint-Leger, A., Poulet, A., Benarroch, D., Magdinier, F., Morere, J., Amiard, S., Verhoeyen, E., Britton, S., Calsou, P., Salles, B., Bizard, A., Nadal, M., Salvati, E., Sabatier, L., Wu, Y., Biroccio, A., Londono-Vallejo, A., Giraud-Panis, M.J., Gilson, E.* (2010) TRF2 and apollo cooperate with topoisomerase 2alpha to protect human telomeres from replicative damage. Cell 142, 230-242 (View in Cell)
2009
Britton, S., Froment, C., Frit, P., Monsarrat, B., Salles, B.*, Calsou, P. (2009) Cell nonhomologous end joining capacity controls SAF-A phosphorylation by DNA-PK in response to DNA double-strand breaks inducers. Cell Cycle 8, 3717-3722 (View in Cell cycle)
Britton, S., Frit, P., Biard, D., Salles, B.*, Calsou, P. (2009) ARTEMIS nuclease facilitates apoptotic chromatin cleavage. Cancer Res 69, 8120-8126 (View in Cancer research)
Wu, P.Y., Frit, P., Meesala, S., Dauvillier, S., Modesti, M., Andres, S.N., Huang, Y., Sekiguchi, J., Calsou, P., Salles, B., Junop, M.S. (2009) Structural and functional interaction between the human DNA repair proteins DNA ligase IV and XRCC4. Mol Cell Biol 29, 3163-3172 (View in Molecular and Cellular Biology)
Britton, S., Salles, B., Calsou, P. (2008) c-MYC protein is degraded in response to UV irradiation. Cell Cycle 7, 63-70 (View in Cell cycle)

