Enveloppes mycobactériennes et cibles thérapeutiques

Responsable d'équipe
L’objectif principal de notre équipe de recherche est la caractérisation de cibles potentielles dans le contexte de l’amélioration de la thérapie antituberculeuse. Notre démarche consiste en l’identification de ces cibles, le décryptage de leurs mécanismes d’action ainsi que leurs rôles dans la physiologie et l’infection. Notre recherche comprend une meilleure compréhension des voies métaboliques et des enzymes impliquées dans la biogenèse des lipides de l’enveloppe cellulaire chez Mycobacterium tuberculosis.
Notre équipe étudie les voies métaboliques des lipides de l'enveloppe mycobactérienne afin de caractériser de nouvelles cibles et concevoir des agents thérapeutiques innovants pour lutter contre la tuberculose dans le contexte de l’antibiorésistance.

La tuberculose (TB), causée par Mycobacterium tuberculosis (Mtb), est l’une des causes majeures de décès dans le monde. L’augmentation de la résistance aux médicaments met en évidence le besoin urgent de développer de nouveaux médicaments antituberculeux. Nous utilisons des approches interdisciplinaires pour découvrir des médicaments potentiels, que ce soit en identifiant et en exploitant des cibles antituberculeuses prometteuses présentant un intérêt pharmaceutique, ou en mettant au point de nouvelles molécules aux modes d’action inexplorés. Notre expertise repose sur des approches complémentaires en biochimie structurale et quantitative des lipides et des glycoconjugués, en microbiologie, en biologie moléculaire, en biotechnologie des protéines et en enzymologie, en développement d’essais enzymatiques et en validation de cibles.
Notre recherche s’articule autour de trois axes :
La caractérisation et inhibition d’enzymes impliquées dans la biogénèse de l’enveloppe mycobactérienne
L’enveloppe mycobactérienne est unique dans le monde bactérien, tant par sa composition riche en composés structurellement exotiques, que par son agencement, avec notamment la présence d’une membrane externe et d’une membrane interne.
Le ciblage des enzymes impliquées dans la biosynthèse de l’enveloppe mycobactérienne présente un intérêt majeur pour la découverte de médicaments antituberculeux. Notre approche est illustrée par l’étude de FAAL32, une acyl-AMP ligase et cible potentielle, impliquée dans la biosynthèse des acides gras à très longue chaîne de l’enveloppe des mycobactéries, appelés acides mycoliques. Pour valider cette protéine en tant que cible pharmacologique, nous avons développé d’un test biochimique qui détecte l’inhibition de FAAL32 et criblé une collection de 1280 médicaments déjà approuvés (Prestwick Chemical Library). Ce criblage a conduit à la découverte d’un composé salicylanilide, le closantel, et de certains de ses dérivés en tant qu’inhibiteurs qui ont une activité importante contre Mtb in vitro. Ces résultats suggèrent que le salicylanilide représente un pharmacophore prometteur pour la conception de nouveaux candidats antituberculeux qui ouvriraient de nouvelles possibilités de ciblage.
La caractérisation structurale et fonctionnelle d’enzymes activant des antibiotiques
De nombreux agents antituberculeux nécessitent une bioactivation, notamment par les Baeyer-Villiger monooxygénases (BVMO). Malgré l’importance émergente de ces enzymes versatiles, les caractéristiques structurales et fonctionnelles de ces BVMO restent mal connues. En combinant une approche in silico et la validation in vitro de l’activité des protéines, nous avons récemment mis en évidence des propriétés structurales et la sélectivité de substrat de ces enzymes. Ces caractéristiques constituent la base moléculaire de la compréhension structure-fonction de la spécificité de ces enzymes et ouvrent la voie à l’élargissement du répertoire des substrats des BVMO, y compris des proantibiotiques.
Nous étudions également les rôles physiologiques des protéines BVMO dans l’adaptation et la résistance de Mtb aux conditions de stress. Nous utilisons des mycobactéries génétiquement modifiées et des caractérisations phénotypiques et lipidomiques pour évaluer les changements induits ainsi que le remodelage de l’enveloppe cellulaire dans des conditions de stress.
L’utilisation de molécules bio-inspirées pour le développement de médicaments antituberculeux
Devant l’émergence croissante de souches de Mtb multi- et extrêmement résistantes aux traitements actuels, il est urgent de développer de nouvelles familles chimiques de médicaments antituberculeux agissant sur des cibles originales. Sur la base d’éléments récents, nous proposons que les lipides bio-inspirés – basés sur des composés isolés de plantes médicinales – représentent une nouvelle classe de molécules antituberculeuses avec de nouveaux mécanismes d’action. En collaboration avec des groupes de chimistes, nous développons une approche interdisciplinaire pour améliorer leur activité contre Mtb mais aussi d’autres mycobactéries pathogènes, en particulier Mycobacterium abscessus, et leurs propriétés pharmacocinétiques tout en diminuant leur cytotoxicité.
Équipe
Chercheurs-Enseignants chercheurs
Anne Lemassu (MCU CE, UPS)
Hedia Marrakchi (CR, CNRS)
Ingénieures
Patricia Constant (IR1, CNRS)
Léa Masson
Doctorants
Martin Campoy
Dimitri Leonelli
Nos projets de recherche (en anglais)
Caractérisation et inhibition d’enzymes impliquées dans la biogénèse de l’enveloppe mycobactérienne
Caractérisation structurale et fonctionnelle d’enzymes activant des antibiotiques
Utilisation de molécules bio-inspirées pour le développement de médicaments antituberculeux
Daffé & Marrakchi (2019) Unraveling the structure of the mycobacterial envelope. Microbiol Spectr
Lanéelle et al. (2021) Lipid and lipoarabinomannan isolation and characterization. Methods Mol Biol
Le et al. (2022) Drug screening approach against mycobacterial fatty acyl-AMP ligase FAAL32 renews the interest of the salicylanilide pharmacophore in the fight against tuberculosis. Bioorg Med Chem
Le et al. (2020) The protein kinase PknB negatively regulates biosynthesis and trafficking of mycolic acids in mycobacteria. J Lipid Res
Tomas et al. (2022) Bioinformatic mining and structure-activity profiling of Baeyer-Villiger monooxygenases from Mycobacterium tuberculosis. mSphere
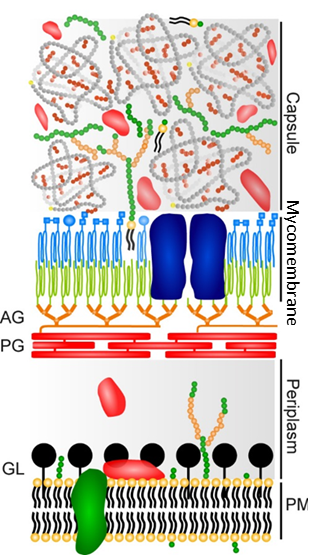
Organization of the mycobacterial envelope. AG, arabinogalactan; PG, peptidoglycan; GL, granular layer; PM, plasma membrane.
Collaborations
International
• European Molecular Biology Laboratory Hamburg, Germany: Matthias Wilmanns
• Institute of Medical Biology – Polish Academy of Sciences, Lodz, Poland : Przemyslaw Plocinski
• University of Padova, Italy : Roberta Provvedi ; Riccardo Manganelli
• Comenius University, Bratislava, Slovak Republic: Jana Korduláková
• Université Libre de Bruxelles : Véronique Fontaine
• SCRIPPS Institute, University of Florida : Luiz Pedro de Carvalho
National
• Institut Pasteur, Paris : Roland Brosch ; Pedro Alzari ; Marco Bellinzoni
• Institut Pasteur Lille/Université de Lille : Alain Baulard ; Nicolas Willand
• MaiAGE, INRAE, Jouy-en-Josas : Gwenaëlle André
• Université Paris XI, Orsay : Nicolas Bayan
• Université Grenoble Alpes : Pierre Verhaeghe
• UMR Infectiologie et Santé Publique, INRAE Tours-Nouzilly : Franck Biet
• Laboratoire Pathogen-Host Interactions, Montpellier : Virginie Molle
• Laboratoire de chimie de coordination LCC, Toulouse: Vania Bernardes-Génisson
• Laboratoire de synthèse et physico-chimie de molécules d’intérêt biologique SPCMIB, Toulouse : Yves Génisson
• Laboratoire Génie Chimique Toulouse : Fatima El Garah
IPBS
• Lionel Mourey
• Odile Schiltz
• Olivier Saurel/Andrew Atkinson
• Sébastien Britton
• Matthieu Chavent
• PICT platform
Funding
Our team is or has been supported in the five past years by grants or fellowships from:
• Agence Nationale de la Recherche (ANR)
• ANRS-Maladies Infectieuses Emergentes (ANRS|MIE)
• MSDAvenir
• Mission pour les Initiatives Transverses et Interdisciplinaires CNRS 80 PRIME et Modélisation du Vivant (Mission pour les Initiatives Transverses et Interdisciplinaires
• Université Paul Sabatier de Toulouse
• Fondation Roland Garrigou
• Partenariat Hubert Curien (Partenariats Hubert Curien (PHC) | Campus France)
The complete list of our publications is available through Pubmed.
Tomas*, N., Leonelli*, D., Campoy, M., Marthey, S., Le, N.-H., Rengel, D., Martin, V., Pál, A., Korduláková, J., Eynard, N., Guillet, V., Mourey, L., Daffé, M., Lemassu*, A., André, G*., Marrakchi*, H. (2022). Bioinformatic Mining and Structure-Activity Profiling of Baeyer-Villiger Monooxygenases from Mycobacterium tuberculosis. MSphere, e0048221 (view)
Le, N.-H., Constant, P., Tranier, S., Nahoum, V., Guillet, V., Maveyraud, L., Daffé, M., Mourey, L., Verhaeghe, P., Marrakchi, H. (2022). Drug screening approach against mycobacterial fatty acyl-AMP ligase FAAL32 renews the interest of the salicylanilide pharmacophore in the fight against tuberculosis. Bioorganic & Medicinal Chemistry, 71, 116938 (view)
Boldrin, F., Cioetto Mazzabò, L., Lanéelle, M.-A., Rindi, L., Segafreddo, G., Lemassu, A., Etienne, G., Conflitti, M., Daffé, M., Garzino Demo, A., Manganelli, R., Marrakchi, H., Provvedi, R. (2022). LysX2 is a Mycobacterium tuberculosis membrane protein with an extracytoplasmic MprF-like domain. BMC Microbiology 22, 85 (view)
Schahl, A., Lemassu, A., Jolibois, F., Réat, V. (2022). Evidence for amylose inclusion complexes with multiple acyl chain lipids using solid-state NMR and theoretical approaches. Carbohydrate Polymers 276, 118749 (view)
Lanéelle, M.-A., Spina, L., Nigou, J., Lemassu, A., Daffé, M. (2021). Lipid and Lipoarabinomannan Isolation and Characterization. Methods in Molecular Biology (Clifton, N.J.) 2314, 109–150 (view)
Le, N.-H., Locard-Paulet, M., Stella, A., Tomas, N., Molle, V., Burlet-Schiltz, O., Daffé, M., Marrakchi, H. (2020). The protein kinase PknB negatively regulates biosynthesis and trafficking of mycolic acids in mycobacteria. Journal of Lipid Research, 61(8), 1180–1191 (view)
Eynard, N., Daffé, M., Brosch, R., Quémard, A. (2020). Discovery of a novel dehydratase of the fatty acid synthase type II critical for ketomycolic acid biosynthesis and virulence of Mycobacterium tuberculosis. Scientific Reports 10, 2112 (view)
Zeng, S., Constant, P., Yang, D., Baulard, A., Lefèvre, P., Daffé, M., Wattiez, R., Fontaine, V. (2019). Cpn60.1 (GroEL1) Contributes to Mycobacterial Crabtree Effect: Implications for Biofilm Formation. Frontiers in Microbiology 10, 1149 (view)
Daffé, M., Marrakchi, H. (2019). Unraveling the Structure of the Mycobacterial Envelope. Microbiology Spectrum 7 (view)
Madacki, J., Laval, F., Grzegorzewicz, A., Lemassu, A., Záhorszká, M., Arand, M., McNeil, M., Daffé, M., Jackson, M., Lanéelle, M.-A., Korduláková, J. (2018). Impact of the epoxide hydrolase EphD on the metabolism of mycolic acids in mycobacteria. The Journal of Biological Chemistry 293, 5172–5184 (view)
Lefebvre, C., Boulon, R., Ducoux, M., Gavalda, S., Laval, F., Jamet, S., Eynard, N., Lemassu, A., Cam, K., Bousquet, M.-P., Bardou, F., Burlet-Schiltz, O., Daffé, M., Quémard, A. (2018). HadD, a novel fatty acid synthase type II protein, is essential for alpha- and epoxy-mycolic acid biosynthesis and mycobacterial fitness. Scientific Reports 8, 6034 (view)
Bannantine, J. P., Etienne, G., Laval, F., Stabel, J. R., Lemassu, A., Daffé, M., Bayles, D. O., Ganneau, C., Bonhomme, F., Branger, M., Cochard, T., Bay, S., Biet, F. (2017). Cell wall peptidolipids of Mycobacterium avium: From genetic prediction to exact structure of a nonribosomal peptide. Molecular Microbiology 105, 525–539 (view)
Bazet Lyonnet, B., Diacovich, L., Gago, G., Spina, L., Bardou, F., Lemassu, A., Quémard, A., Gramajo, H. (2017). Functional reconstitution of the Mycobacterium tuberculosis long-chain acyl-CoA carboxylase from multiple acyl-CoA subunits. The FEBS Journal 284, 1110–1125 (view)
Issa, H., Huc-Claustre, E., Reddad, T., Bonadé Bottino, N., Tropis, M., Houssin, C., Daffé, M., Bayan, N., Dautin, N. (2017). Click-chemistry approach to study mycoloylated proteins: Evidence for PorB and PorC porins mycoloylation in Corynebacterium glutamicum. PloS One 12, e0171955 (view)
Carel, C., Marcoux, J., Réat, V., Parra, J., Latgé, G., Laval, F., Demange, P., Burlet-Schiltz, O., Milon, A., Daffé, M., Tropis, M. G., Renault, M. A. M. (2017). Identification of specific posttranslational O-mycoloylations mediating protein targeting to the mycomembrane. Proceedings of the National Academy of Sciences of the United States of America 114, 4231–4236 (view)
Chiaradia, L., Lefebvre, C., Parra, J., Marcoux, J., Burlet-Schiltz, O., Etienne, G., Tropis, M., Daffé, M. (2017). Dissecting the mycobacterial cell envelope and defining the composition of the native mycomembrane. Scientific Reports 7, 12807 (view)
Daffé, M. (2015). The cell envelope of tubercle bacilli. Tuberculosis (Edinburgh, Scotland), 95 Suppl 1, S155-158 (view)
Boritsch, E. C., Frigui, W., Cascioferro, A., Malaga, W., Etienne, G., Laval, F., Pawlik, A., Le Chevalier, F., Orgeur, M., Ma, L., Bouchier, C., Stinear, T. P., Supply, P., Majlessi, L., Daffé, M., Guilhot, C., Brosch, R. (2016). Pks5-recombination-mediated surface remodelling in Mycobacterium tuberculosis emergence. Nature Microbiology 1, 15019 (view)
Guillet, V., Galandrin, S., Maveyraud, L., Ladevèze, S., Mariaule, V., Bon, C., Eynard, N., Daffé, M., Marrakchi, H., Mourey, L. (2016). Insight into Structure-Function Relationships and Inhibition of the Fatty Acyl-AMP Ligase (FadD32) Orthologs from Mycobacteria. The Journal of Biological Chemistry 291, 7973–7989 (view)
Le, N.-H., Molle, V., Eynard, N., Miras, M., Stella, A., Bardou, F., Galandrin, S., Guillet, V., André-Leroux, G., Bellinzoni, M., Alzari, P., Mourey, L., Burlet-Schiltz, O., Daffé, M., Marrakchi, H. (2016). Ser/Thr Phosphorylation Regulates the Fatty Acyl-AMP Ligase Activity of FadD32, an Essential Enzyme in Mycolic Acid Biosynthesis. The Journal of Biological Chemistry 291, 22793–22805 (view)
Burbaud, S., Laval, F., Lemassu, A., Daffé, M., Guilhot, C., Chalut, C. (2016). Trehalose Polyphleates Are Produced by a Glycolipid Biosynthetic Pathway Conserved across Phylogenetically Distant Mycobacteria. Cell Chemical Biology 23, 278–289 (view)
Slama, N., Jamet, S., Frigui, W., Pawlik, A., Bottai, D., Laval, F., Constant, P., Lemassu, A., Cam, K., Daffé, M., Brosch, R., Eynard, N., Quémard, A. (2016). The changes in mycolic acid structures caused by hadC mutation have a dramatic effect on the virulence of Mycobacterium tuberculosis. Molecular Microbiology 99, 794–807 (view)
Jamet, S., Quentin, Y., Coudray, C., Texier, P., Laval, F., Daffé, M., Fichant, G., Cam, K. (2015). Evolution of Mycolic Acid Biosynthesis Genes and Their Regulation during Starvation in Mycobacterium tuberculosis. Journal of Bacteriology 197, 3797–3811 (view)
Jamet, S., Slama, N., Domingues, J., Laval, F., Texier, P., Eynard, N., Quémard, A., Peixoto, A., Lemassu, A., Daffé, M., Cam, K. (2015). The Non-Essential Mycolic Acid Biosynthesis Genes hadA and hadC Contribute to the Physiology and Fitness of Mycobacterium smegmatis. PloS One 10, e0145883 (view)
Ehebauer, M. T., Zimmermann, M., Jakobi, A. J., Noens, E. E., Laubitz, D., Cichocki, B., Marrakchi, H., Lanéelle, M.-A., Daffé, M., Sachse, C., Dziembowski, A., Sauer, U., Wilmanns, M. (2015). Characterization of the mycobacterial acyl-CoA carboxylase holo complexes reveals their functional expansion into amino acid catabolism. PLoS Pathogens 11, e1004623 (view)
Daffé, M., Crick, D. C., Jackson, M. (2014). Genetics of Capsular Polysaccharides and Cell Envelope (Glyco)lipids. Microbiology Spectrum 2, MGM2-0021–2013 (view)
Marrakchi, H., Lanéelle, M.-A., Daffé, M. (2014). Mycolic acids: Structures, biosynthesis, and beyond. Chemistry & Biology 21, 67–85 (view)
Moura-Alves, P., Faé, K., Houthuys, E., Dorhoi, A., Kreuchwig, A., Furkert, J., Barison, N., Diehl, A., Munder, A., Constant, P., Skrahina, T., Guhlich-Bornhof, U., Klemm, M., Koehler, A.-B., Bandermann, S., Goosmann, C., Mollenkopf, H.-J., Hurwitz, R., Brinkmann, V., Fillatreau, S., Daffé, M., … Kaufmann, S. H. E. (2014). AhR sensing of bacterial pigments regulates antibacterial defence. Nature 512, 387–392 (view)
Rienksma, R. A., Suarez-Diez, M., Spina, L., Schaap, P. J., Martins dos Santos, V. A. P. (2014). Systems-level modeling of mycobacterial metabolism for the identification of new (multi-)drug targets. Seminars in Immunology 26, 610–622 (view)
Gavalda, S., Bardou, F., Laval, F., Bon, C., Malaga, W., Chalut, C., Guilhot, C., Mourey, L., Daffé, M., Quémard, A. (2014). The polyketide synthase Pks13 catalyzes a novel mechanism of lipid transfer in mycobacteria. Chemistry & Biology 21, 1660–1669 (view)
Galandrin, S., Guillet, V., Rane, R. S., Léger, M., N, R., Eynard, N., Das, K., Balganesh, T. S., Mourey, L., Daffé, M., Marrakchi, H. (2013). Assay development for identifying inhibitors of the mycobacterial FadD32 activity. Journal of Biomolecular Screening 18, 576–587 (view)
Gavalda, S., Léger, M., van der Rest, B., Stella, A., Bardou, F., Montrozier, H., Chalut, C., Burlet-Schiltz, O., Marrakchi, H., Daffé, M., Quémard, A. (2009). The Pks13/FadD32 crosstalk for the biosynthesis of mycolic acids in Mycobacterium tuberculosis. The Journal of Biological Chemistry 284, 19255–19264 (view)
Lefebvre, C., Frigui, W., Slama, N., Lauzeral-Vizcaino, F., Constant, P., Lemassu, A., Parish, T., Léger, M., Gavalda, S., Guillet, V., van der Rest, B., Slama, N., Montrozier, H., Mourey, L., Quémard, A., Daffé, M., Marrakchi, H. (2009). The dual function of the Mycobacterium tuberculosis FadD32 required for mycolic acid biosynthesis. Chemistry & Biology 16, 510–519 (view)
• Pascaline Bories-Lafont, post-doctoral fellow, CHU Québec-Université Laval, Quebec
• Nicolas Tomas, post-doctoral fellow, TWB, France
• Laura Chiaradia, Analytical Engineer, Centre Hospitalier Territorial, New Caledonia
• Nguyen-Hung Le, post-doctoral fellow, Marseille, France
• Richard Boulon, post-doctoral fellow, INRS institute, Quebec
• Cyril Lefebvre, Unit Manager, Dubernet Rhône, France
• Mathieu Leger, Quality Manager, iNOVie, France
• Julien Vaubourgeix, Lecturer, Imperial College, London, UK
• Tounkang Sambou, Unit Manager, Senegal
• Nawel Slama, Project Manager, TWB, France
• Emilie Huc, Research Scientist, Evotec, France
• Premkumar Dinadayala, Research Scientist, Evotec, France
• Emmanuelle Sacco, Clinical Research Project Manager, CHU, France
• Jean-Clément Mars, Post-doctoral fellow, Laval, Québec
• Brahim Saffiedine, Product Manager, Senegal
• Lucie Spina, Analytical Chemist, Clariant Active Ingredients, France
• Sabine Gavalda, Biophysics Engineer, Carbios, France
• Ségolène Galandrin, Project Manager, CHU, France

